Ricercatori dell’IZSVe hanno identificato due genotipi di virus influenzali finora sconosciuti in Italia, denominati Novel 1 e Novel 2, in popolazioni di suini. La scoperta è avvenuta nell’ambito di uno studio di sorveglianza in allevamenti suini del Nord-Est, pubblicato sulla rivista Frontiers in Microbiology. Il genotipo Novel 1, appartenente al sottotipo H1avN2, è stato isolato in Friuli Venezia Giulia, mentre il genotipo Novel 2, riconducibile al sottotipo H1pdmN2, è stato identificato in Veneto. Lo studio prosegue una linea di ricerca che già nel 2018 aveva permesso di identificare altri genotipi virali.
L’influenza suina è una malattia respiratoria dei suini diffusa in tutto il mondo, con importanti implicazioni per la salute animale e umana.
“Il suino è una specie estremamente interessante per lo studio dei ceppi influenzali, dal momento che è suscettibile all’infezione di ceppi influenzali di diversa origine, suina, umana e aviare” spiega Lara Cavicchio, biotecnologa ricercatrice del Laboratorio di genomica e trascrittomica virale dell’Istituto Zooprofilattico Sperimentale delle Venezie (IZSVe), prima autrice dell’articolo. “Per questa ragione il suino rappresenta un mixing vessel, una sorta di ‘miscelatore’ dove i virus influenzali di diversa origine possono andare incontro ad eventi di riassortimento, cioè si scambiano pezzi di materiale genetico, creando nuove varianti, potenzialmente pericolose per l’uomo.”
Un esempio tangibile delle conseguenze negative di questo meccanismo si è avuto con la prima pandemia del XXI secolo. Era il 2009 e a scatenarla fu un virus influenzale H1N1 originato dal riassortimento fra ceppi virali suini, umani e aviari. La sorveglianza è dunque fondamentale per monitorare la circolazione e l’evoluzione di questi virus, al fine di studiare il loro potenziale zoonosico ed eventualmente pandemico.
L’analisi genetica evidenzia la circolazione inter aziendale del virus
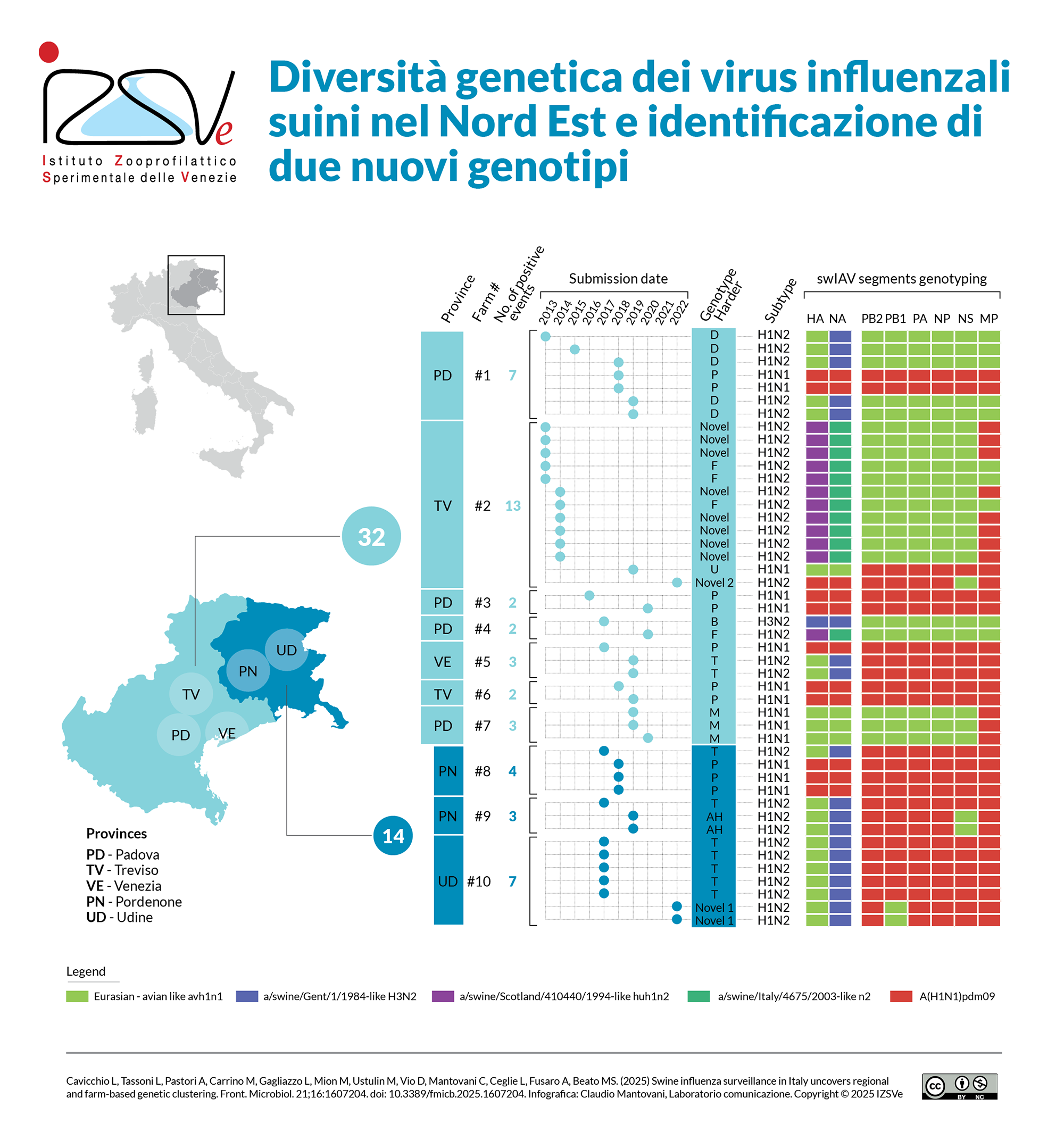
Ricercatori dell’IZSVe hanno identificato due genotipi del virus dell’influenza suina finora sconosciuti in Italia, denominati Novel 1 e Novel 2. La scoperta è avvenuta nell’ambito di uno studio di sorveglianza in allevamenti suini del Nord-Est: grazie alla collaborazione tra veterinari aziendali e ricercatori, sono stati raccolti tra il 2013 e il 2022 oltre 3.000 campioni in Veneto, Friuli Venezia Giulia e Trentino-Alto Adige da animali sintomatici. La presenza di cluster genetici condivisi tra allevamenti, spesso localizzati in aree geografiche contigue o appartenenti alla stessa rete produttiva, indica fenomeni di diffusione inter-aziendale. In alcuni casi, le sequenze virali mostravano elevata similarità con ceppi umani, coerente con possibili eventi di trasmissione inter-specifica.
Grazie alla collaborazione tra veterinari aziendali e ricercatori, sono stati raccolti tra il 2013 e il 2022 oltre 3.000 campioni in Veneto, Friuli Venezia Giulia e Trentino-Alto Adige da animali sintomatici: circa il 19% è risultato positivo a influenza suina. La maggior parte dei campioni positivi proviene dalle aree a più alta densità suinicola, nelle province di Padova, Verona, Treviso, Pordenone e Udine.
Con la caratterizzazione genetica dei due nuovi genotipi “italiani” Novel 1 e Novel 2, ad oggi i genotipi noti nel Nord est sono dodici, di cui dieci già descritti precedentemente (A, B, D, F, M, P, T, U, AH e Novel2013).
L’analisi filogenetica, effettuata per tracciare la storia evolutiva dei virus e le loro “parentele”, ha evidenziato la presenza di distinti cluster genetici, costituiti da virus strettamente correlati in allevamenti diversi, e ha rilevato una positività ricorrente a virus influenzali suini in numerose aziende nel corso del periodo di sorveglianza. Questi dati suggeriscono sia la persistenza intra-aziendale di specifici ceppi, sia reintroduzioni ricorrenti legate alle dinamiche di filiera.
La presenza di cluster genetici condivisi tra allevamenti, spesso localizzati in aree geografiche contigue o appartenenti alla stessa rete produttiva, indica fenomeni di diffusione inter-aziendale. In alcuni casi, le sequenze virali mostravano elevata similarità con ceppi umani, coerente con possibili eventi di trasmissione interspecifica.
Una sorveglianza continua per prevenire rischi di spillover nell’uomo
I risultati ottenuti evidenziano la complessità dell’epidemiologia dei virus influenzali suini e confermano il ruolo del suino come serbatoio e mixing vessel. L’elevata eterogeneità genetica, la persistenza di ceppi all’interno degli stessi allevamenti e la presenza di cluster condivisi tra diverse aziende sono indicativi di una circolazione virale continua e interconnessa, determinata sia da fenomeni di persistenza intra-aziendale sia da reintroduzioni lungo le filiere produttive.
Questi dati sottolineano la necessità di implementare una sorveglianza continua e integrata, in cui la caratterizzazione genetica dei virus sia correlata a informazioni gestionali ed epidemiologiche. Tale approccio è fondamentale per monitorare l’evoluzione antigenica dei ceppi circolanti, guidare strategie vaccinali efficaci e ridurre il rischio di spillover verso l’uomo.
Leggi l’articolo scientifico su Frontiers in Microbiology »