Questo report descrive le caratteristiche genetiche di un totale di 1104 campioni di SARS-CoV-2, di cui 1068 prelevati tra novembre 2020 e maggio 2021 e 36 campioni raccolti tra marzo e ottobre 2020.
Su mandato regionale l’Istituto Zooprofilattico Sperimentale delle Venezie (IZSVe) sta monitorando le caratteristiche genetiche e la variabilità dei ceppi di SARS-CoV-2 presenti in Veneto. L’emergere di mutazioni nel genoma di agenti virali ad RNA come SARS-CoV-2 è un evento naturale ed atteso. Cambiamenti nella trasmissibilità del virus, nella gravità della malattia, nella capacità del virus di sfuggire all’immunità acquisita (post-infezione o vaccinazione) e ai test diagnostici in uso: questi sono gli elementi cruciali che definiscono le dinamiche di interazione di SARS-CoV-2 con la popolazione ospite. Sequenziare il genoma di un virus significa poter riconoscere l’emergere di varianti virali che possono modificare l’andamento e l’impatto dell’epidemia. Le mutazioni più interessanti sono a livello della proteina Spike del virus data l’importanza che questa riveste per il legame con i recettori cellulari e perché verso di essa sono rivolti i principali anticorpi che danno la protezione verso l’infezione e le forme cliniche.
Si ringraziano tutti coloro che hanno condiviso le sequenze nel database pubblico GISAID e le ULSS 1, ULSS 2, ULSS 3, ULSS 4, ULSS 5, ULSS 6, ULSS 7, ULSS 8, ULSS 9, U.O.C Microbiologia e virologia dell’Azienda ospedaliera-Università di Padova e dell’Azienda ospedaliera universitaria integrata di Verona che hanno inviato i campioni oggetto di questo report.
In evidenza
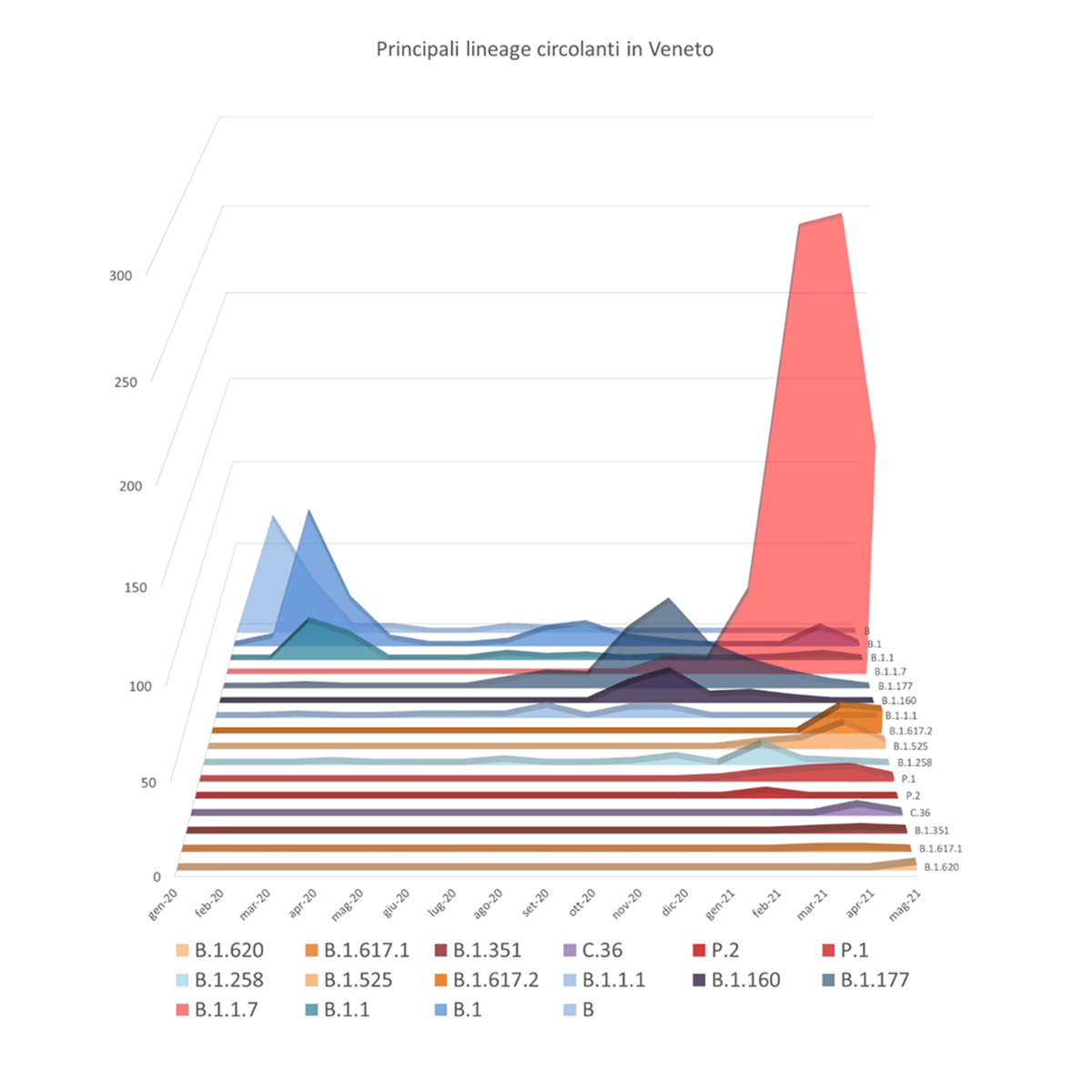
- Identificati 28 lineage in Veneto dal mese di gennaio 2021. Nel mese di maggio 2021 è stata individuata la circolazione di 9 lineage distinti, di cui quattro Variant of Concern (VOC): B.1.1.7 (variante inglese), B.1.617.2 (variante indiana), B.1.351 (variante sudafricana), e P.1 (variante brasiliana).
- Il risultato della sorveglianza “Prevalenza delle varianti VOC (Variant Of Concern) in Italia: lineage B.1.1.7, P.1 e lineage B.1.351, e altre varianti (Variant Of Interest, VOI) tra cui lineage P.2 e lineage B.1.525” del 18 maggio, coordinata da ISS, ha confermato una prevalenza del 94,2% della variante inglese VOC-202012/01 (lineage B.1.1.7) in Veneto. Le varianti B.1.617.2 (variante indiana) e P.1 (variante brasiliana) sono state identificate con una frequenza inferiore al 2%. La variante nigeriana B.1.525 e la variante sudafricana B.1.351 non sono state individuate nel corso di questa sorveglianza.
- Sono state caratterizzate per la prima volta in Veneto le varianti B.1.620 (inclusa tra le VOI da ECDC) e C.36 (inclusa tra le varianti da monitorare da ECDC), ed è stata identificata per la prima volta in Italia la variante B.1.499.
- La variante B.1.620 si caratterizza per un elevato numero di mutazioni nella proteina Spike, molte delle quali in comune con altre VOC. Tali mutazioni potrebbero contribuire a rendere il virus meno suscettibile alla risposta anticorpale.
Limiti dello studio
Il numero di campioni sequenziati ad oggi da IZSVe è limitato rispetto al numero di casi positivi in Veneto e fornisce solo un’istantanea parziale delle possibili varianti circolanti nel territorio.
Descrizione dei nuovi campioni sequenziati in questo report
Nell’ambito del DGR 1424 del 21/10/2020 è stato ottenuto il genoma completo di un totale di 1104 campioni, di cui 1068 prelevati tra novembre 2020 e maggio 2021 e 36 campioni raccolti tra marzo e ottobre 2020.
Resoconto delle varianti rilevate in Veneto
Figura 1. Grafico dei principali lineage identificati in Veneto da gennaio 2020 a maggio 2021.
Tabella 1. Lista delle varianti identificate in Veneto a partire dal mese di gennaio 2021. In grassetto le varianti indicate da ECDC e WHO come variants of concern (VOC) e variants of interest (VOI) (ECDC, WHO). I dati riportati fanno riferimento solamente ai virus di cui si dispongono sequenze del genoma completo sequenziati da IZSVe o disponibili in GISAID.
| lineage | VOC/VOI | gen-21 | feb-21 | mar-21 | apr-21 | mag-21 | tot |
|---|---|---|---|---|---|---|---|
| B.1.1.7 | VOC | 7 | 49 | 257 | 263 | 135 | 711 |
| B.1.177 | 25 | 15 | 8 | 3 | 0 | 51 | |
| B.1.617.2 | VOC | 0 | 0 | 0 | 15 | 13 | 28 |
| B.1.525 | VOI | 0 | 3 | 5 | 14 | 4 | 26 |
| P.1 | VOC | 1 | 4 | 6 | 7 | 2 | 20 |
| B.1.258 | 0 | 11 | 2 | 1 | 0 | 14 | |
| B.1.258.17 | 0 | 4 | 7 | 2 | 0 | 13 | |
| B.1 | 0 | 0 | 0 | 11 | 1 | 12 | |
| B.1.160 | 4 | 5 | 2 | 0 | 0 | 11 | |
| C.36 | 0 | 0 | 0 | 5 | 1 | 6 | |
| B.1.2 | 0 | 0 | 1 | 4 | 0 | 5 | |
| B.1.351 | VOC | 0 | 0 | 1 | 2 | 1 | 4 |
| B.1.177.75 | 0 | 0 | 4 | 0 | 0 | 4 | |
| B.1.1 | 0 | 0 | 1 | 3 | 0 | 4 | |
| P.2 | VOI | 0 | 3 | 0 | 0 | 0 | 3 |
| B.1.620 | VOI | 0 | 0 | 0 | 0 | 3 | 3 |
| B.1.1.318 | 0 | 0 | 3 | 0 | 0 | 3 | |
| B.1.1.29 | 0 | 1 | 2 | 0 | 0 | 3 | |
| I.1 | 0 | 2 | 0 | 0 | 0 | 2 | |
| B.1.617.1 | VOC | 0 | 0 | 1 | 1 | 0 | 2 |
| B.1.499 | 0 | 0 | 0 | 0 | 1 | 1 | |
| B.1.221 | 1 | 0 | 0 | 0 | 0 | 1 | |
| B.1.177.10 | 0 | 1 | 0 | 0 | 0 | 1 | |
| B.1.1.420 | 0 | 0 | 1 | 0 | 0 | 1 | |
| B.1.1.136 | 0 | 1 | 0 | 0 | 0 | 1 | |
| B.1.1.10 | 1 | 0 | 0 | 0 | 0 | 1 | |
| AH.3 | 0 | 1 | 0 | 0 | 0 | 1 | |
| A | 1 | 0 | 0 | 0 | 0 | 1 |
Variante Indiana B.1.617
La variante B.1.617, identificata per la prima volta in India a fine 2020, si suddivide in tre gruppi genetici – B.1.617.1, B.1.617.2, B.1.617.3 – i quali si distinguono per un diverso profilo di mutazioni. La variante B.1.617 è stata designata come VOC (Variant of Concern) l’11 aprile 2021 dal WHO.
Situazione epidemiologica in Veneto
In base ai risultati della sorveglianza in Veneto si evidenzia solo un moderato aumento dei casi legati alla variante B.1.617 nel mese di maggio (1.5%) rispetto al mese precedente (0.6%).
Complessivamente nella regione sono stati identificati 2 casi della variante B.1.617.1 nel mese di aprile a Vicenza e 31 casi (28 confermati con il sequenziamento del genoma completo) della variante B.1.617.2 tra aprile e maggio in campioni provenienti dalle province di Treviso, Venezia, Padova, Verona, Vicenza. Molti di questi casi sono stati riscontati in soggetti di rientro dall’India e loro familiari, ma si ritiene possibile una crescente diffusione di questa variante nel territorio regionale. In particolare, i due casi identificati nel corso della sorveglianza ISS del 18 maggio non riportano nessun viaggio o contatto con persone di rientro dall’estero.
Caratteristiche della variante B.1.617.2
In base ai dati preliminari la variante B.1.617.2 sembra essere caratterizzata da una maggiore trasmissibilità rispetto alla variante B.1.1.7.
Inoltre, un recente studio pubblicato da Public Health England il 22 maggio 2021 (Bernal et al., preprint) riporta che l’efficacia del vaccino (dopo la seconda dose) nel prevenire l’insorgenza dei sintomi in seguito all’infezione con la variante B.1.617.2 è comparabile all’efficacia riportata per la variante B.1.1.7. In particolare:
- dopo due settimane dalla seconda dose il vaccino Pfizer-BioNTech ha un’efficacia dell’88% nel prevenire la sintomatologia causata dall’infezione con la variante B.1.617.2 e del 93% in caso di infezione con variante B.1.1.7;
- due dosi del vaccino AstraZeneca hanno un’efficacia del 60% nel prevenire la sintomatologia causata dall’infezione con la variante B.1.617.2 e del 66% in caso di infezione B.1.1.7.
Sebbene rassicuranti, tali dati si basano sull’analisi di un numero relativamente ridotto di casi e necessitano di essere confermati nelle prossime settimane.
Variante B.1.620
La variante B.1.620 (identificata anche come variante camerunense) è stata individuata in diversi paesi europei, inclusa l’Italia, e dell’Africa Centrale, da cui si ipotizza essersi originata (Dudas et al., preprint). Il 24 maggio l’ECDC ha incluso tale variante tra le VOI (Variants of Interest).
Situazione epidemiologica in Veneto
Ad oggi in Veneto segnaliamo cinque casi dovuti a questa variante, tre dei quali identificati nel corso della sorveglianza ISS del 18 maggio in campioni provenienti da Padova e Belluno.
Caratteristiche della variante B.1.620
Tale variante si caratterizza per un elevato numero di mutazioni e delezioni nella proteina Spike (P26S, 69-70del, V126A, 144del, 241-243del, H245Y, S477N, E484K, D614G, P681H, T1027I e D1118H), alcune delle quali (E484K, S477N le delezioni HV69del, Y144del e LLA241/243del) presenti anche in molte VOC. In particolare tale variante contiene nel dominio N-terminale e nel sito recettoriale della proteina Spike una serie di mutazioni che sono state precedentemente associate ad una riduzione della capacità neutralizzante da parte di specifici anticorpi monoclonali o sieri di soggetti guariti dall’infezione (Dudas et al., preprint). Ulteriori studi sono necessari per meglio comprendere le caratteristiche di tale variante.
Variante C.36
La variante C.36, diffusa prevalentemente in Egitto, è stata individuata in diversi altri Paesi, tra cui Regno Unito, USA, Germania e Svizzera. In Italia è stata identificata per la prima volta a luglio 2020, in Abruzzo. In data 7 maggio 2021 l’ECDC ha inserito questa variante tra quelle da monitorare, per la presenza nella proteina Spike di mutazioni che potrebbero destare particolare interesse (L452R, D614G, Q677H).
Situazione epidemiologica in Veneto
In Veneto segnaliamo otto casi dovuti a questa variante: uno identificato nel corso della sorveglianza ISS del 18 maggio (Verona), e cinque identificati in campioni prelevati nelle province di Padova e Treviso nel periodo aprile-maggio 2021.
Caratteristiche della variante C.36
Questa variante si caratterizza per le seguenti mutazioni nella proteina Spike: S12F, 69-70del, W152R, R346S, L452R, D614G, Q677H, A899S, alcune delle quali (L452R e Q677H) presenti anche in alcune VOC. La mutazione L452R, pur non entrando direttamente in contatto con il recettore umano ACE2, sembra essere associata a una maggiore stabilità dell’interazione tra il recettore e la proteina Spike, e quindi a una possibile maggiore infettività (Chen et al., 2020, Deng et al., preprint). Questa mutazione, inoltre, sembra correlare con una ridotta capacità neutralizzante da parte di alcuni anticorpi monoclonali (Li et al., 2020). Ulteriori studi sono necessari per approfondire le caratteristiche di questa variante.
Risultato della sorveglianza ISS – 18 maggio 2021
Tabella 2. Varianti rilevate nella sorveglianza “Prevalenza delle varianti VOC (Variant Of Concern) in Italia: lineage B.1.1.7, P.1 e lineage B.1.351, e altre varianti (Variant Of Interest, VOI) tra cui lineage P.2 e lineage B.1.525” del 18 maggio.
| Lineage | N. sequenze | Mutazioni nella proteina Spike | |
|---|---|---|---|
| B.1.1.7 (variante inglese) | VOC | 129 | |
| B.1.499 | 1 | D614G, A626S | |
| B.1.620 | 2 | P26S, V126A, L242F, A243T, H245Y, S247I, S477N, E484K, D614G, P681H, T1027I, D1118H, del69-70-, del144 | |
| B.1.620 | 1 | P26S, V126A, H245Y, S254F, S477N, E484K, D614G, P681H, T1027I, D1118H, del69-70, del144-, del241-243 | |
| B.1.525 (variante nigeriana) | VOI | 0 | |
| P.1 (variante brasiliana) | VOC | 1 | |
| B.1.351 (variante sudafricana) | VOC | 0 | |
| B.1.617.2 (variante indiana) | VOC | 2 | |
| C.36/B.1.1.1.36 | 1 | S12F, W152R, R346S, L452R, D614G, Q677H, A899S, del69-70 | |
| TOTALE | 137 |
Si segnala che in alcuni campioni del lineage B.1.1.7 abbiamo osservato la comparsa di mutazioni nel sito di legame al recettore della proteina Spike: A344S (N=1), V367F (N=2), V382L (N=4), P384L (N=1), K417N (N=1), L452R (N=1), A475V (N=2), S477I (N=1), T478K (N=1), P479S (N=4), E484K (N=3), F490S (N=1).
Referenze
- Bernal J.L. et al. (2021). Effectiveness of COVID-19 vaccines against the B.1.617.2 variant. merdRxiv (preprint)
- Chen J., Wang R., Wang M., Wei G.W. (2020). Mutations Strengthened SARS-CoV-2 Infectivity. Journal of Molecular Biology, 432 (19), 5212-5226
- Dudas G. et al. (2021). Travel-driven emergence and spread of SARS-CoV-2 lineage B.1.620 with multiple VOC-like mutations and deletions in Europe. merdRxiv (preprint)
- Deng X et al. (2021). Transmission, infectivity, and antibody neutralization of an emerging SARS-CoV-2 variant in California carrying a L452R spike protein mutation. merdRxiv (preprint)
- Li Q et al. (2020). The Impact of Mutations in SARS-CoV-2 Spike on Viral Infectivity and Antigenicity. Cell, 182(5), 1284-1294.e9