Questo report descrive le caratteristiche genetiche di campioni di SARS-CoV-2 identificati in Veneto tra il 2 novembre 2020 e il 24 agosto 2021.
Su mandato regionale l’Istituto Zooprofilattico Sperimentale delle Venezie (IZSVe) sta monitorando le caratteristiche genetiche e la variabilità dei ceppi di SARS-CoV-2 presenti in Veneto. L’emergere di mutazioni nel genoma di agenti virali ad RNA come SARS-CoV-2 è un evento naturale ed atteso. Cambiamenti nella trasmissibilità del virus, nella gravità della malattia, nella capacità del virus di sfuggire all’immunità acquisita (post-infezione o vaccinazione) e ai test diagnostici in uso: questi sono gli elementi cruciali che definiscono le dinamiche di interazione di SARS-CoV-2 con la popolazione ospite. Sequenziare il genoma di un virus significa poter riconoscere l’emergere di varianti virali che possono modificare l’andamento e l’impatto dell’epidemia. Le mutazioni più interessanti sono a livello della proteina Spike del virus data l’importanza che questa riveste per il legame con i recettori cellulari e perché verso di essa sono rivolti i principali anticorpi che danno la protezione verso l’infezione e le forme cliniche.
Si ringraziano tutti coloro che hanno condiviso le sequenze nel database pubblico GISAID, le ULSS 1, ULSS 2, ULSS 3, ULSS 4, ULSS 5, ULSS 6, ULSS 7, ULSS 8, ULSS 9, U.O.C Microbiologia e virologia dell’Azienda ospedaliera-università di Padova e dell’Azienda ospedaliera universitaria integrata di Verona che hanno inviato i campioni oggetto di questo report e l’UOSD Genetica e Citogenetica – Azienda ULSS 3 Serenissima per aver contribuito alla generazione delle sequenze.
In evidenza
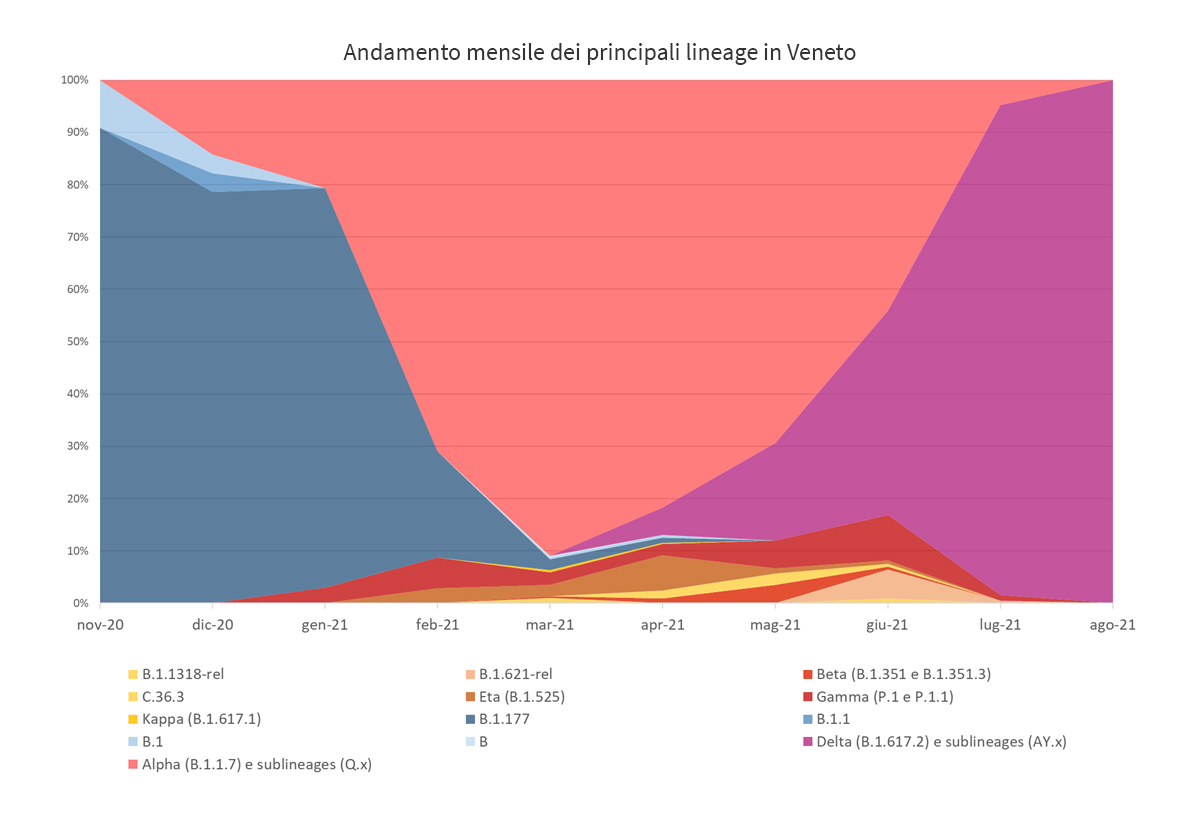
- Il 100% dei campioni caratterizzati in Veneto nel mese di agosto 2021 appartengono alla variante Delta. L’estesa circolazione della variante è risultata nell’emergere di molteplici lineaggi. In Veneto si segnala l’identificazione di 10 lineage. In particolare, il 46% delle sequenze ottenute appartiene al lineage B.1.617.2, mentre il restante 54% ricade all’interno di 9 sublineage (AY.x).
- Il risultato della sorveglianza “Stima della prevalenza delle varianti VOC (Variant Of Concern) in Italia: lineage B.1.1.7, P.1, B.1.617.(1,2 o 3) e B.1.351, e altre varianti del virus SARS-CoV-2” del 24 agosto, coordinata da ISS, ha evidenziato un ulteriore aumento della prevalenza in Veneto della variante Delta rispetto al mese precedente, passando dal 97,2% di luglio al 100% di agosto, e la scomparsa delle altre varianti precedentemente circolanti.
- Si evidenzia una diversificazione della variante Delta in diversi sublineage e l’acquisizione di mutazioni nella proteina Spike, alcune delle quali identificate in altre VOC.
Limiti dello studio
Il numero di campioni del Veneto sequenziati ad oggi è limitato rispetto al numero di casi positivi nella regione e fornisce solo un’istantanea parziale delle possibili varianti circolanti nel territorio.
Descrizione dei nuovi campioni sequenziati in questo report
Nell’ambito del DGR 1424 del 21/10/2020 e successiva nota regionale (prot. n. 304579) è stato ottenuto il genoma completo di un totale di 3.033 campioni, di cui 2.997 prelevati tra novembre 2020 e luglio 2021 e 36 campioni raccolti tra marzo e ottobre 2020.
Resoconto delle varianti rilevate in Veneto
Figura 1. Grafico dei principali lineage identificati in Veneto da gennaio 2020 ad agosto 2021. Nell’asse delle ordinate è riportata la frequenza delle principali varianti identificate.
Variante Delta – B.1.617.2
La variante Delta (lineage B.1.617.2), segnalata per la prima volta in India a fine 2020, è stata designata come VOC (Variant of Concern) dall’WHO l’11 aprile 2021. L’estensiva circolazione di tale variante ha portato alla sua diversificazioni in molteplici sublineages (es. B.1.617.2.1, B.1.617.2.2, B.1.617.2.3 ecc.) identificati per semplicità come AY.x (es. AY.1 alias B.1.617.2.1, AY.2 alias B.1.617.2.2, AY.3 alias B.1.617.2.3 ecc.).
Situazione epidemiologica in Veneto
Complessivamente nella regione sono stati sequenziati 1582 campioni appartenenti alla variante Delta tra aprile e agosto 2021, in campioni provenienti da tutte le province del Veneto.
In base ai risultati della sorveglianza ISS, in Veneto si evidenzia un aumento della prevalenza della variante Delta nel mese di agosto (100%) rispetto al mese precedente (97.22%). In generale, il 100% dei campioni caratterizzati nel mese di agosto appartengono a questa variante, suddivisi all’interno di 10 differenti lineages elencati in tabella 2. I lineage B.1.617.2, AY.4 e AY.12 sono quelli più diffusi.
Tabella 2. Frequenza dei diversi lineage della variante Delta identificati in Veneto nel mese di agosto 2021
| Lineage | Numerosità | Frequenza |
|---|---|---|
| B.1.617.2 | 212 | 46,2% |
| AY.4 | 128 | 27,9% |
| AY.12 | 55 | 12,0% |
| AY.6 | 32 | 7,0% |
| AY.7 | 12 | 2,6% |
| AY.9 | 8 | 1,7% |
| AY.7.2 | 5 | 1,1% |
| AY.5 | 3 | 0,7% |
| AY.14 | 3 | 0,7% |
| AY.25 | 1 | 0,2% |
Analisi delle mutazioni di interesse – variante Delta
In questo report sono stati sequenziati un totale di 888 campioni. Tutte le sequenze di agosto appartengono alla variante delta e su queste è stata condotta un’ulteriore analisi per ricercare la presenza di mutazioni aggiuntive a quelle caratterizzanti la variante stessa e i suoi sublineages. Sono state riportate in particolare le mutazioni nella proteina Spike tipiche di altre VOC, VOI e VUM, quelle note per avere particolari effetti su trasmissibilità e patogenicità o le mutazioni all’interno del receptor binding domain (RBD).
- L5F identificata in 3 sequenze: tipica della VOI Iota (B.1.526); probabilmente associata all’ingresso della proteina Spike nel reticolo endoplasmatico, oltre a essere un sito riconosciuto dall’antigene leucocitario umano
- T95I identificata in 56 sequenze: tipica delle VOI Iota (B.1.526), B.1.1.318 e B.1.621; gli effetti di questa mutazione sono tuttora sconosciuti
- Y144del identificata in 1 sequenza: tipica della VOC Alpha (B.1.1.7) e di altre VOI (Eta, B.1.1.318); associata a un ridotto riconoscimento da parte di alcuni anticorpi
- A243del identificata in 1 sequenza: tipica della variante Beta (B.1.351); nel dominio N-terminale della Spike, non se ne conosce ancora l’impatto a livello biologico
- Q677H identificata in 3 sequenze: tipica delle VOI Eta (B.1.525) e C36.3; si trova nel sito di legame della furina per il clivaggio S1/S2, e potrebbe pertanto avere effetti sull’ingresso del virus nella cellula ospite
- H1101Y identificata in 1 sequenza: tipica della variante Theta (P.3); si ipotizza che possa aumentare la stabilità del monomero della proteina Spike
Si evidenziano inoltre le seguenti mutazioni riscontrate in singole sequenze che ricadono nel RBD: A348S, N354K, N370S, G446V, S477I.
Risultato della sorveglianza ISS – 24 agosto 2021
Si riportano sinteticamente i risultati ottenuti nell’ultima sorveglianza straordinaria nazionale sulle varianti di SARS-CoV-2 coordinata dall’ISS sui campioni positivi COVID-19 notificati il 24 agosto 2021, per un totale di 160 campioni analizzati.
I virus sequenziati appartengono tutti alla variante Delta e si possono suddividere nel lineage B.1.617.2 e nei suoi relativi sublineage AY.x come riportato in tabella 3.
Tabella 3. Frequenza dei diversi lineage della variante Delta identificati in Veneto ad agosto
| Lineage | Numerosità | Frequenza |
|---|---|---|
| B.1.617.2 | 59 | 36,9% |
| AY.4 | 54 | 33,8% |
| AY.12 | 30 | 18,8% |
| AY.9 | 5 | 3,1% |
| AY.6 | 4 | 2,5% |
| AY.14 | 3 | 1,9% |
| AY.7 | 2 | 1,3% |
| AY.7.2 | 2 | 1,3% |
| AY.25 | 1 | 0,6% |
La variante delta è stata identificata in tutte le province. La sua prevalenza nel territorio regionale è cresciuta di 2,78 punti percentuali rispetto alla sorveglianza precedente (luglio), passando dal 97,22% al 100%, confermandosi come la variante prevalente.
Referenze
- European Centre for Disease Prevention and Control (ECDC). SARS-CoV-2 variants of concern as of 22 July 2021
- World Health Organization (WHO). Tracking SARS-CoV-2 variants
- GISAID – https://www.gisaid.org/
- Virological – virological.org
- Lubinski B, Tang T, Daniel S, Jaimes JA, Whittaker GR. (2021). Functional evaluation of proteolytic activation for the SARS-CoV-2 variant B.1.1.7: role of the P681H mutation. bioRxiv [Preprint]. 2021 Apr 8:2021.04.06.438731. doi: 10.1101/2021.04.06.438731.
- Timmers, L.F.S.M., Peixoto, J.V., Ducati, R.G. et al. (2021). SARS-CoV-2 mutations in Brazil: from genomics to putative clinical conditions. Sci Rep 11, 11998 (2021). doi: 10.1038/s41598-021-91585-6
- Nagy A, Basiouni S, Parvin R, Hafez HM, Shehata AA. Evolutionary insights into the furin cleavage sites of SARS-CoV-2 variants from humans and animals. Arch Virol. 2021 Sep;166(9):2541-2549. doi: 10.1007/s00705-021-05166-z



![«Emergenze sanitarie ai tempi di internet». Imparare dalla storia per affrontare le emergenze sanitarie [Libro]](https://www.izsvenezie.it/wp-content/uploads/2024/07/emergenze-sanitarie-tempi-internet-500x383.jpg)


