Di seguito i risultati ottenuti nell’ambito dell’attività sorveglianza coordinata dall’Istituto Superiore di Sanità (ISS) “Stima della prevalenza delle varianti VOC (Variant Of Concern) e di altre varianti di SARS-CoV-2 in Italia” sui campioni positivi per COVID-19 notificati in Veneto il 10 gennaio 2023.
È stato ottenuto il genoma completo di 158 campioni inviati da 13 diversi laboratori distribuiti nella regione. Il 100% dei campioni analizzati appartiene alla variante Omicron.
Varianti identificate
All’interno della variante Omicron si distinguono cinque lineage principali BA.1, BA.2, BA.3, BA.4 e BA.5 e diversi sublineage. BA.2, BA.4 e BA.5 e relativi sublineage sono classificati da ECDC come VOC (Variant of Concern). Nel corso della sorveglianza del 10 gennaio in Veneto sono stati identificati i seguenti lineage:
| Variante | % |
|---|---|
| BA.5 e sublineage (BA.5*) | 86,1% |
| BA.2 e sublineage (BA.2*) | 8,9% |
| BA.4 e sublineage (BA.4*) | 0,6% |
| Ricombinanti | 4,4% |
Nel corso di questa sorveglianza le varianti BA.4 e BA.5 complessivamente hanno raggiunto una prevalenza del 86,7% (86,1% delle quali sono BA.5), in calo rispetto alla sorveglianza del mese di dicembre (89,8%). Il lineage BA.2 è calato di 1 punto percentuale rispetto al mese di dicembre (10,2%) ed ha raggiunto la prevalenza del 8.9%. Sono stati identificati 7 campioni ricombinanti di cui tre appartenenti alla variante XBB (XBB.1 e XBB.1.5) e tre appartenenti alle varianti XBF, XBG e XBJ, due delle quali (XBG e XBJ) identificate in Veneto per la prima volta. Il numero di virus ricombinanti risulta in aumento rispetto alla sorveglianza di dicembre, in cui sono stati rilevati 3 campioni ricombinanti, tutti appartenenti alla variante XBB.
All’interno di questa sorveglianza si segnalano i seguenti lineage tutti identificati dal WHO come VUM (Variant Under Monitoring):
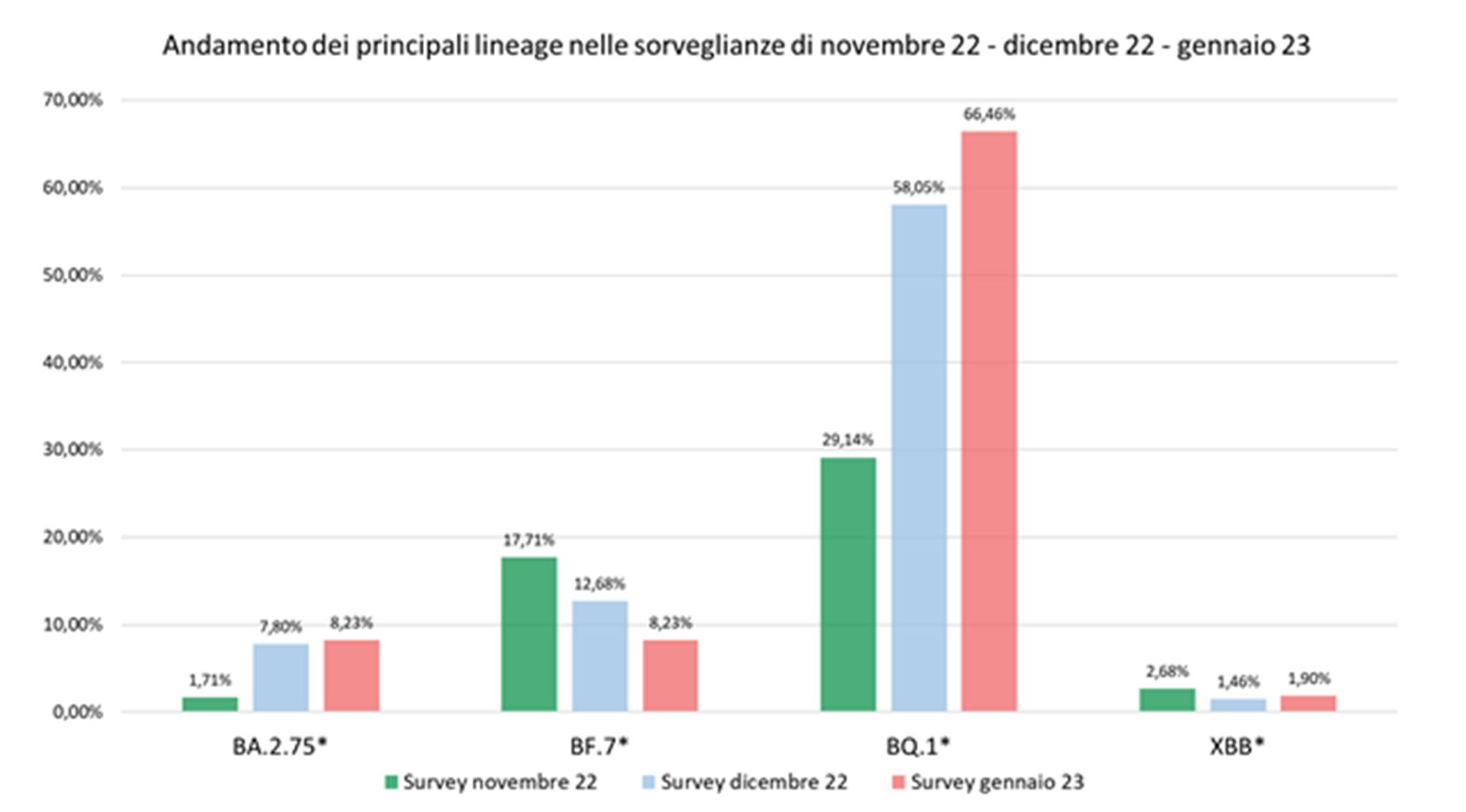
- BQ.1 e sublineage (Cerberus), appartenete alla variante BA.5, si riconferma il lineage più rappresentato in Veneto con una prevalenza del 66.5%. Rispetto alla precedente sorveglianza la sua frequenza è aumentata di 8,5 punti percentuali.
- Ricombinante XBB e sublineage è stato identificato per la prima volta in Veneto nel corso del mese di novembre. XBB è un ricombinante dei sublineage BA.2, BJ.1 (BA.2.10.1.1) e BM.1.1.1 (BA.2.75.3.1.1.1) (punto di ricombinazione a livello del Receptor Binding Domain della proteina Spike). In questa sorveglianza si segnala la presenza di due sequenze appartenenti al sublineage XBB.1.5 (Kraken), una nella provincia di Vicenza e una nella provincia di Verona. Nell’ultimo update dell’ECDC del 9 gennaio 2023 si stima che il sublineage XBB.1.5 abbia un notevole vantaggio di crescita rispetto ai lineage circolanti in Nord America (109%) e Europa (113%). Tale variante desta preoccupazione negli Stati Uniti dove è arrivata a rappresentare, nella settimana terminata il 14 gennaio 2023, il 43% circa delle sequenze analizzate. Il vantaggio di crescita di questa variante sembra possa essere una conseguenza dell’elevata capacità di evasione della risposta immunitaria (già dimostrata da XBB), combinata a una maggiore affinità di legame per il recettore umano ACE2 (hACE2) rispetto alle sottovarianti BQ.1.1 e XBB/XBB.1. Tale affinità di legame deriva della presenza della mutazione S486P nella proteina Spike che differenzia XBB.1.5 da XBB e XBB.1. L’elevata affinità per hACE2 potrebbe consentire a XBB.1.5 di acquisire in futuro ulteriori mutazioni responsabili di un aumento di fenomeni di immune escape, e per tale ragione il suo andamento va attentamente monitorato a livello globale.
Sulla base delle sequenze depositate in GISAID al 9 gennaio 2023, tale variante è stata rilevata prevalentemente negli Stati Uniti (4111 sequenze) e nel Regno Unito (202 sequenze) ma anche in diversi paesi Europei tra i quali l’Italia (5 sequenze di cui 2 individuate in provincia di Venezia a fine dicembre da parte dell’UOSD Genetica e Citogenetica dell’Ospedale dell’Angelo di Mestre).
- BM, BN, CH, CJ (BA.2.75.X) sono sublineage rispettivamente di BA.2.75.3, di BA.2.75.5, BA.2.75.3.4.1.1, BA.2.75.3.4.1.1.1.1. Rispetto al lineage BA.2, la variante BA.2.75.X presenta 9 mutazioni aggiuntive a livello della Spike (S:K147E, S:W152R, S:F157L, S:I210V, S:G257S, S:D339H, S:G446S, S:N460K, reversione Q493R). La percentuale di tutti i sublineage appartenenti a BA.2.75.X (Centaurus) ha raggiunto l’8.2%, in leggero aumento rispetto al mese di dicembre (7.8%).
In particolare il lineage CH.1.1 (BA.2.75.3.4.1.1.1.1), individuato per la prima volta in Veneto a fine novembre in provincia di Padova, è stato rilevato nel 3.8% delle sequenze analizzate in questo monitoraggio. La mutazione S:P681R, responsabile di un aumento di fusogenicità e patogenicità nei virus che la possiedono, non è stata rilevata. - CM.7 (BA.2.3.20.7) è stato identificato in un campione della provincia di Rovigo. Rispetto al lineage BA.2, la variante BA.2.3.20 presenta 9 mutazioni aggiuntive a livello della Spike (S:M153T, S:N164K, S:H245N, S:G257D, S:K444R, S:N450D, S:L452M, S:N460K, S:E484R). Tale variante è stata segnalata per la prima volta in Veneto lo scorso settembre e il suo rilevamento si conferma per ora sporadico.
- BF.7 e sublineage è stato rilevato nell’8,2% delle sequenze analizzate in questa sorveglianza, in calo rispetto alla sorveglianza di dicembre (12,7%)
Di seguito si può osservare il cambiamento percentuale dei lineage BA.2.75*, BF.7* E BQ.1* rispetto alle due sorveglianze precedenti del 8 novembre e del 13 dicembre.
Segnaliamo inoltre l’identificazione di virus ricombinanti XBF, XBG, XBJ, derivati tutti da eventi di ricombinazione avvenuti tra il lineage BA.2 e il lineage BA.5, in particolare:
- XBF è un ricombinante dei lineage BA.5.2.3 e CJ.1 (BA.2.75.3.1.1.1.1) con potenziale sito di ricombinazione prima della posizione 9866 (ORF1a).
- XBJ è un ricombinante dei lineage BA.2.3.20 e BA.5.2 con potenziale punto di ricombinazione tra la proteina Spike e la ORF3a (23015-25809).
- XBG è un ricombinante dei lineage BA.2.76 e BA.5.2 con potenziale sito di ricombinazione all’interno della proteina Spike (22601 e 22915).
Tali ricombinanti sono già stati individuati sporadicamente in Italia. In Veneto, la variante XBF è stata identificata nella provincia di Verona a dicembre da parte dell’UOC Microbiologia, AOUI Verona, mentre si tratta della prima individuazione delle varianti XBG e XBJ.
Distribuzione sul territorio
I lineage si ripartiscono nelle diverse province del Veneto come segue:
| Provincia | BA.2* | BA.4* | BA.5* | Ricombinanti |
|---|---|---|---|---|
| Belluno | 25,0% | – | 75,0% | – |
| Padova | 15,6% | – | 75,0% | 9,4% |
| Rovigo | 7,7% | – | 92,3% | – |
| Treviso | – | – | 90,0% | 10,0% |
| Venezia | 6,3% | 3,1% | 90,6% | – |
| Verona | 7,1% | – | 89,3% | 3,6% |
| Vicenza | 8,0% | – | 88,0% | 4,0% |
In particolare la distribuzione percentuale dei lineage VUM BQ.1, BF.7, BA.2.75 (BM, BN, CH, CJ) e XBB nelle varie province è la seguente:
| Provincia | BQ.1 e sublineage | BF.7 e sublineage | BA.2.75 e sublineage | XBB e sublineage |
|---|---|---|---|---|
| Belluno | 62,5% | – | 25,0% | – |
| Padova | 50,0% | 9,4% | 15,6% | – |
| Rovigo | 84,6% | – | – | – |
| Treviso | 60,0% | 15,0% | – | 5,0% |
| Venezia | 71,9% | 3,1% | 6,3% | – |
| Verona | 67,9% | 14,3% | 7,1% | 3,6% |
| Vicenza | 76,0% | 8,0% | 8,0% | 4,0% |
Ringraziamenti
Si ringraziano l’UOSD Genetica e Citogenetica – Azienda ULSS 3 Serenissima per aver contribuito alla generazione delle sequenze e le ULSS 1, ULSS 2, ULSS4, ULSS 5, ULSS 6, ULSS 7, ULSS 8, ULSS 9, le U.O.C Microbiologia e virologia dell’Azienda ospedaliera-Università di Padova e dell’Azienda ospedaliera universitaria integrata di Verona che hanno selezionato e inviato i campioni oggetto della sorveglianza.